HSvu
Разработчик:
Panalytical
Описание
The HSvu-app (HighScore View) displays all kinds of X-ray diffraction scans in various formats.
Additionally, it shows and reports all details from an X-ray diffraction analysis performed by the HighScore analysis software from PANalytical (proprietary .HPF format).
Experience the power and ease-of-use of HighScore with HSvu:
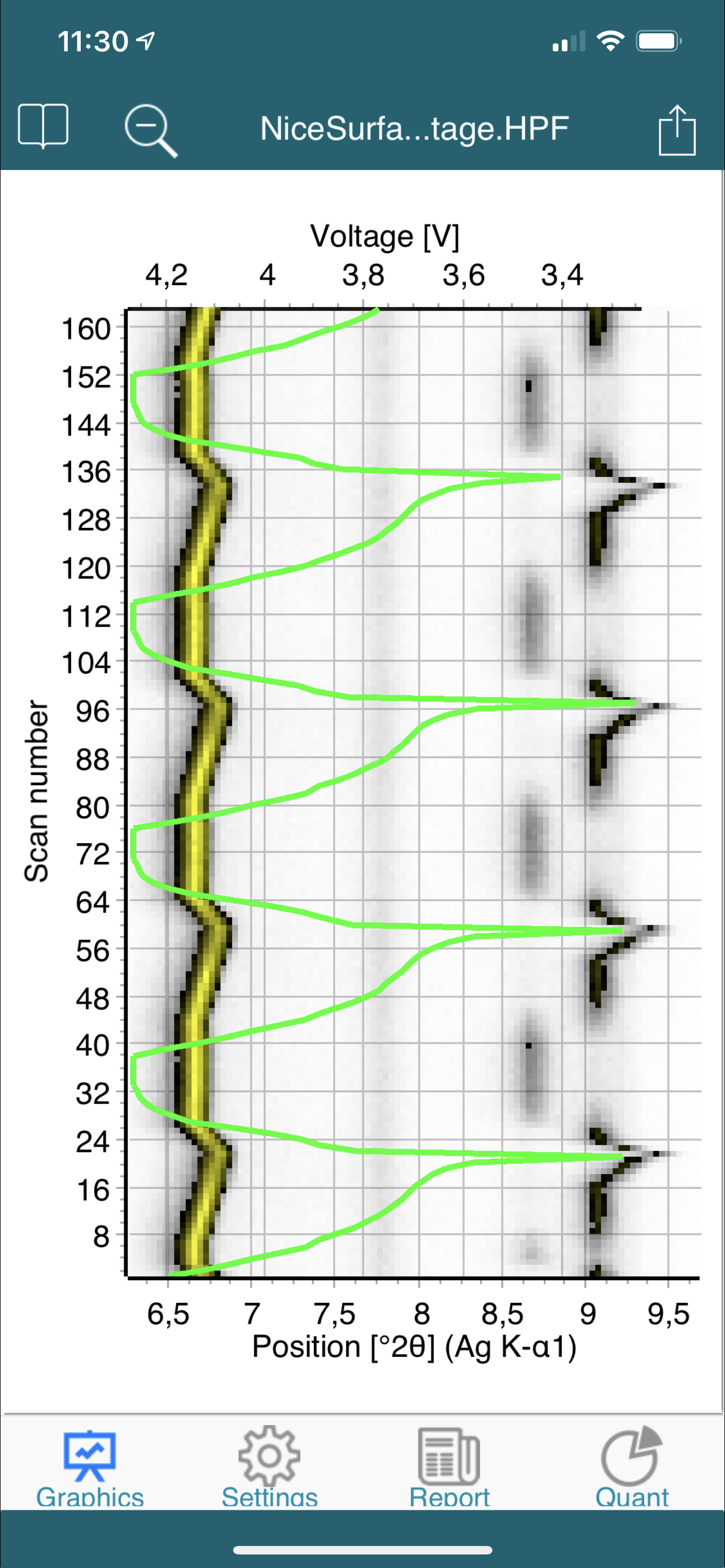
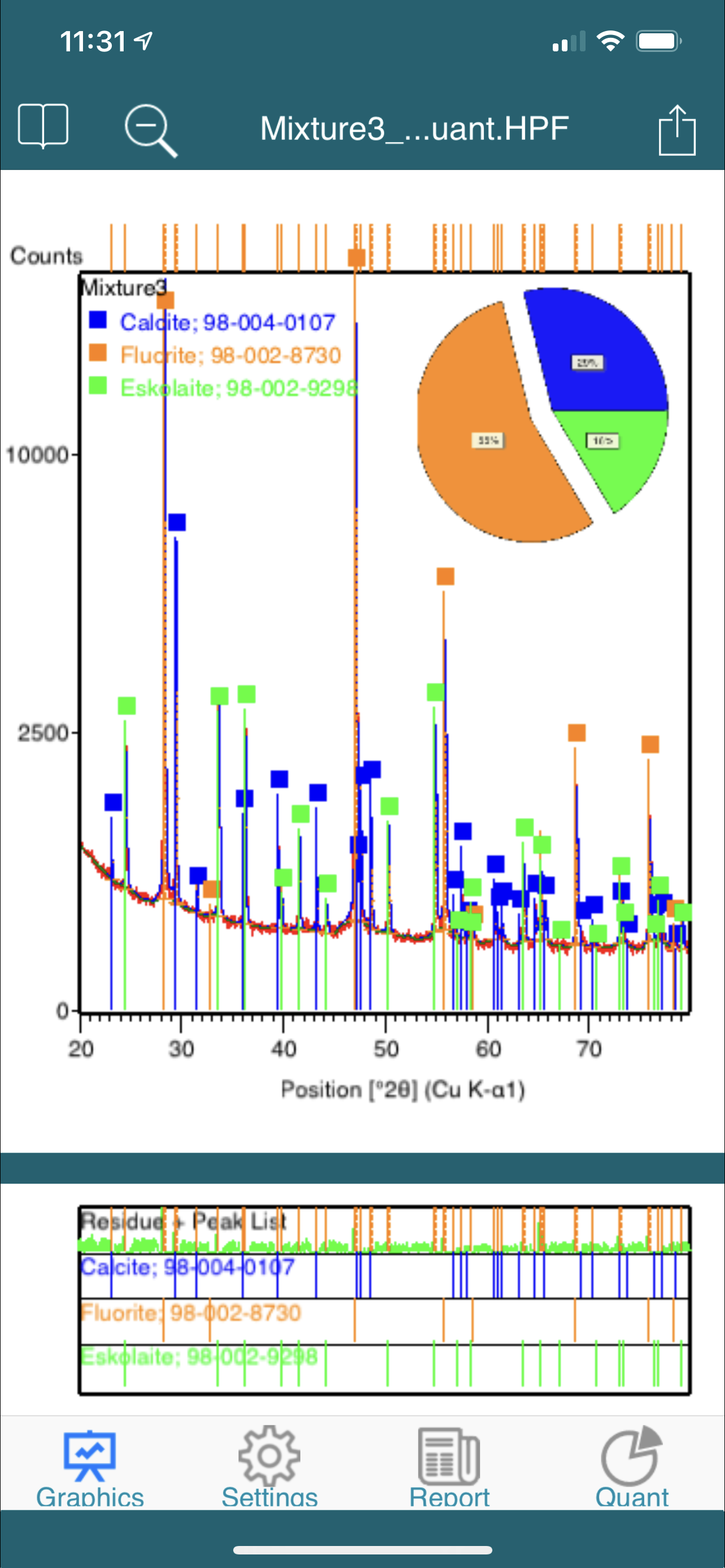
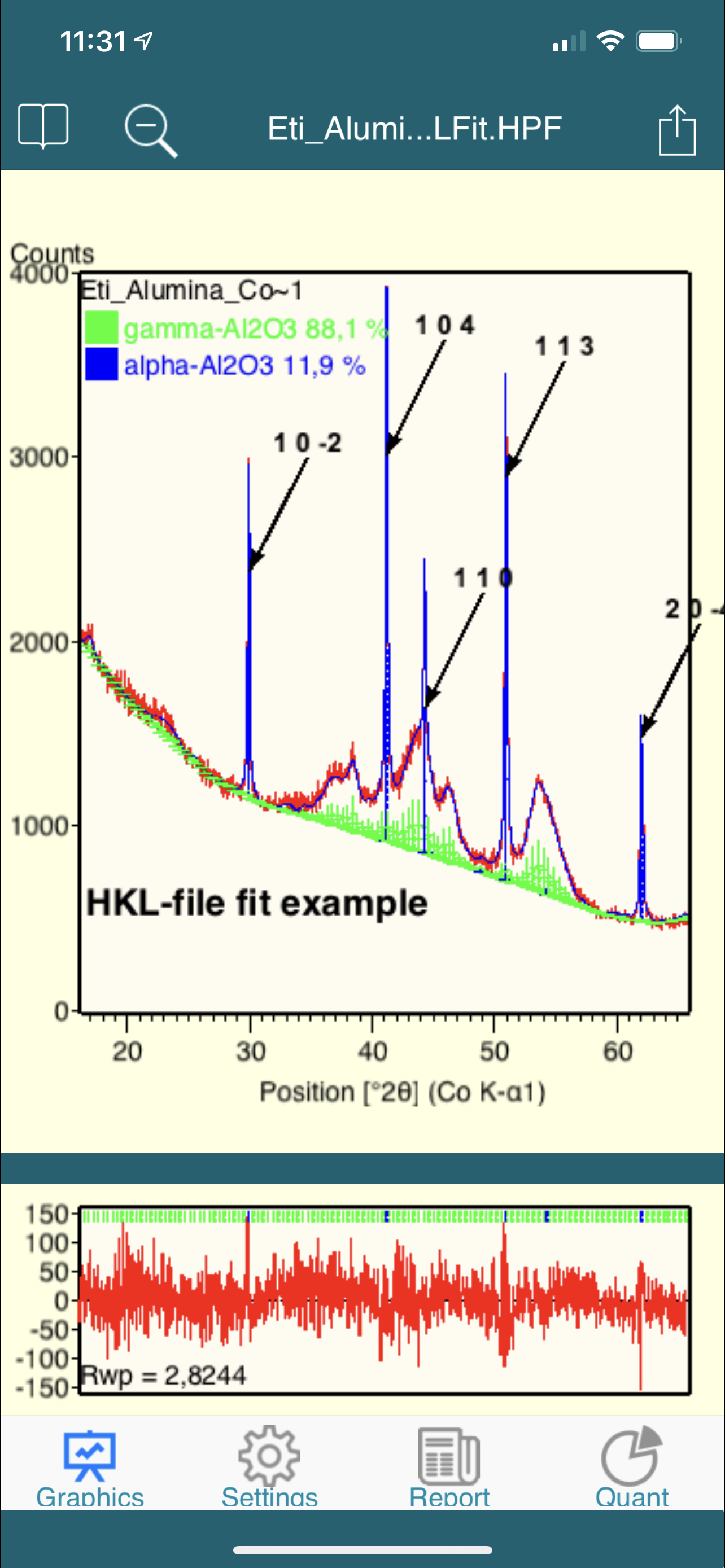
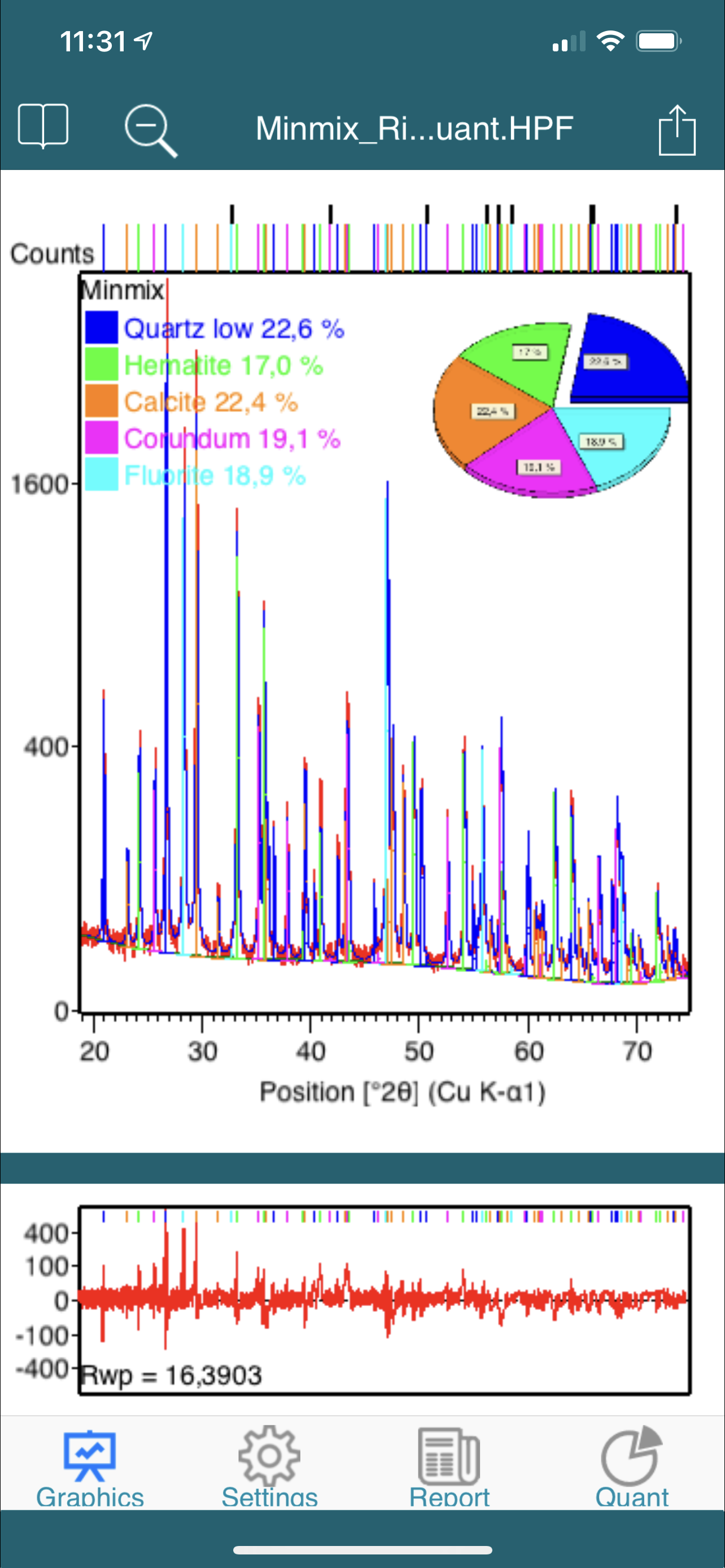
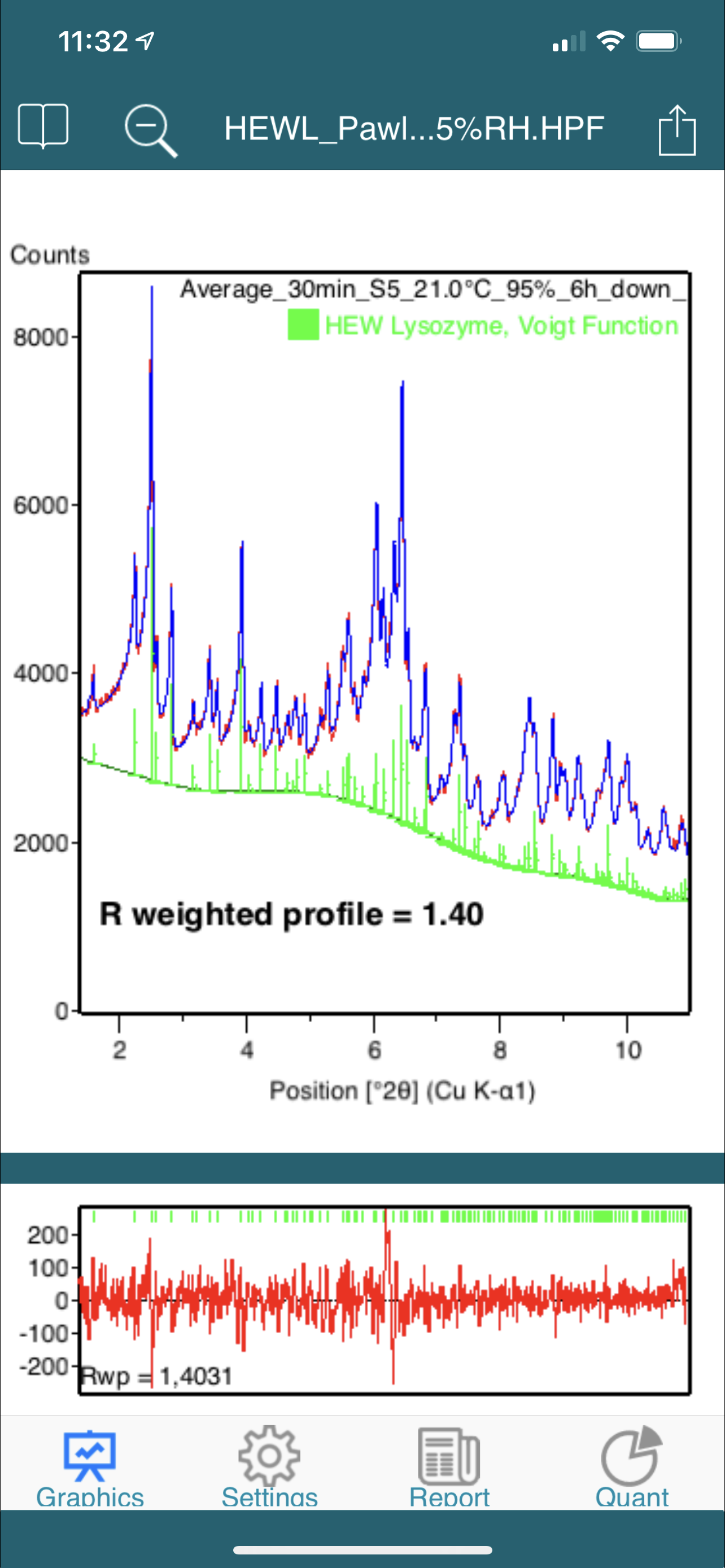
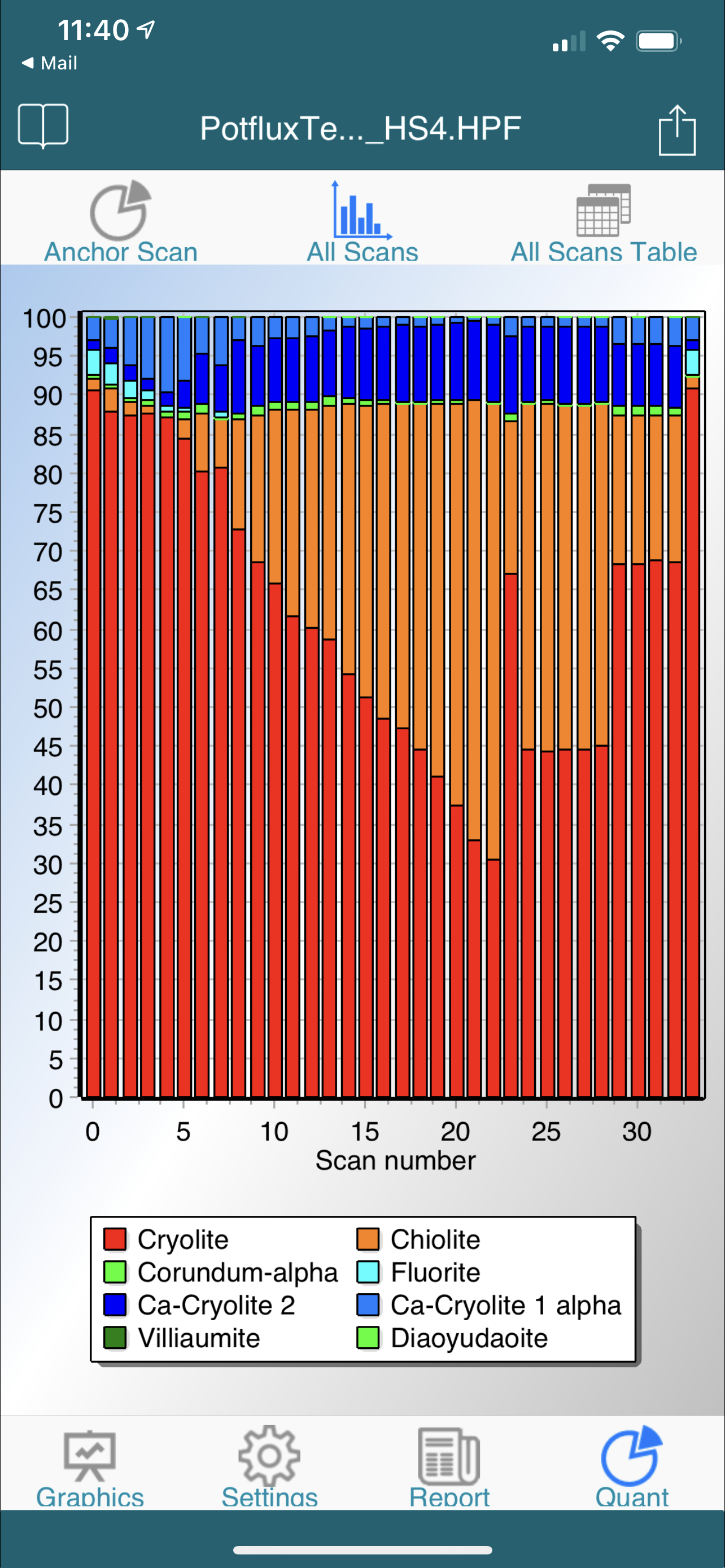
- use six different graphical views for references, single and multiple scans, quantifications and 3-dimensional graphs
- pan and zoom-in for details like you do with HighScore
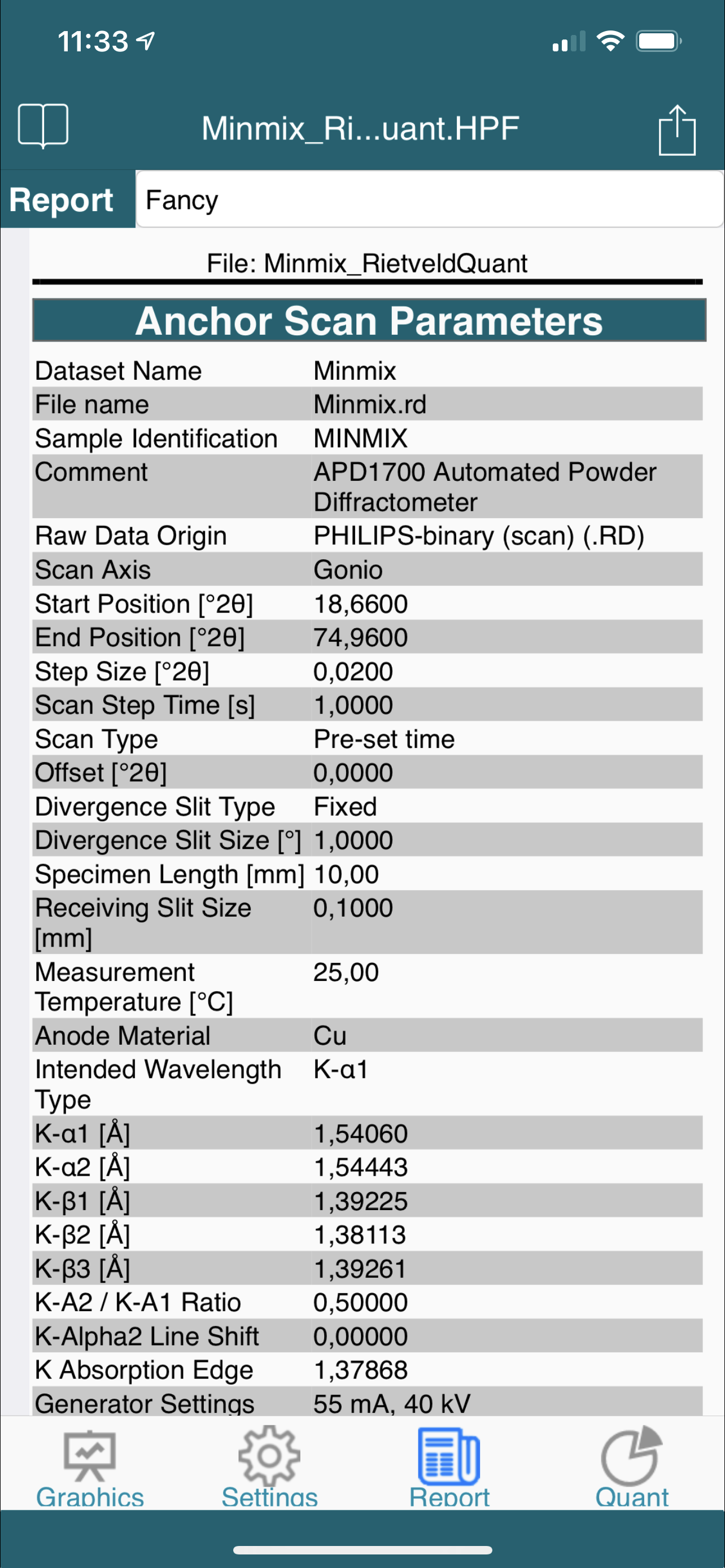
- get a complete overview on measurement conditions, scans, peaks, profiles and crystal structure data
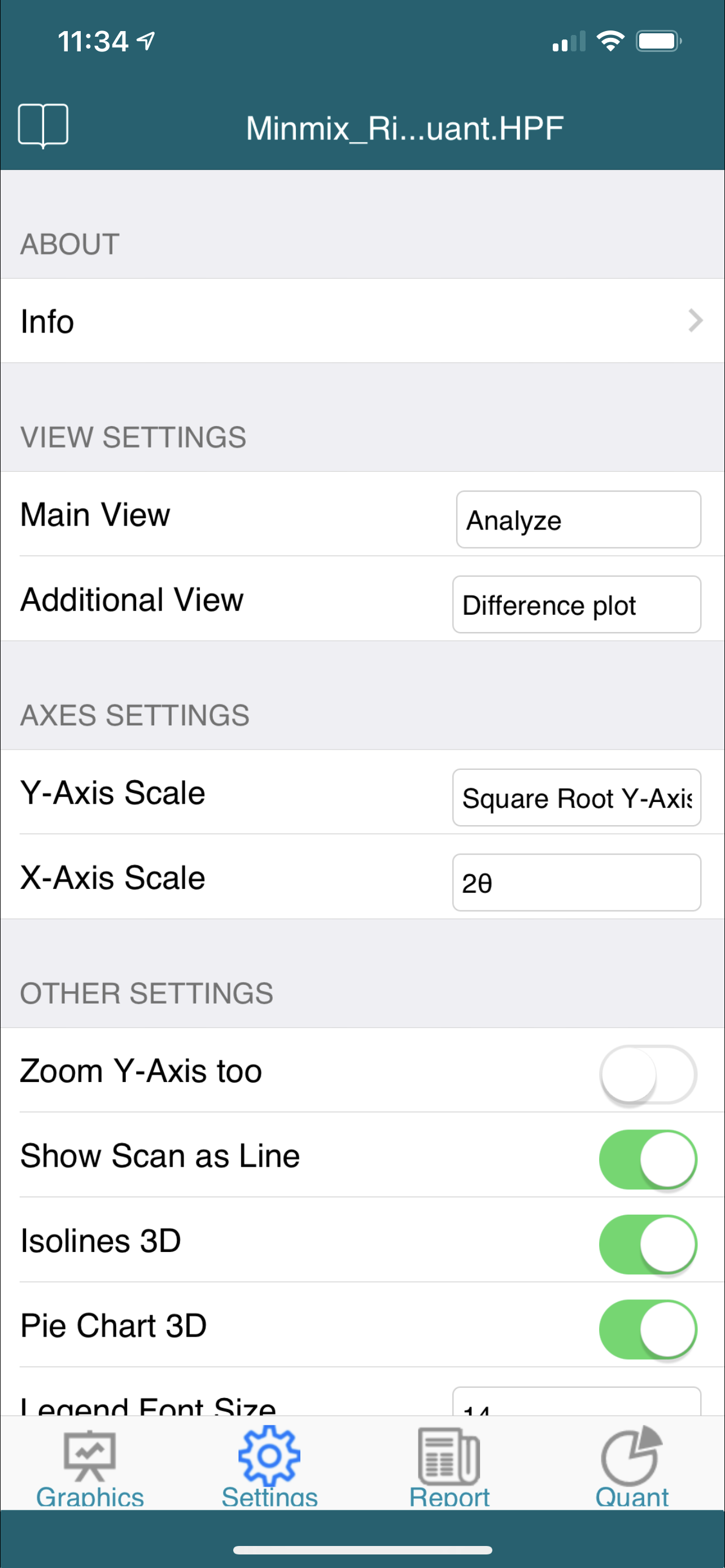
- switch easily between graphics and textual information
- unravel the parameters used in a diffraction analysis and its history
- create reports covering either pieces or the complete diffraction document
HSvu is a display or viewing app.
It shows existing data, but it doesn't perform an analysis nor does it change data.
The following file formats are supported:
- PANalytical .HPF (proprietary HighScore document)
- PANalytical scans ( .XRDML)
- PANalytical ( .2Dint), ASCII format integrated from a 2D area detector
- ASD Inc. binary spectrum ( .ASD)
- Philips binary scans ( .RD), ( .SD), ( .FP), ASCCI ( .UDF), ASCII( .LIS)
- Bruker binary scans ( .RAW) versions 1, 2, 3 and 4
- ASCII scans ( .LST), ( .UXD), ( .XDD)
- Crystallographic information file: ( .CIF)
- General ASCII scan X-Y(-Z) table ( .ASC)
- DBW-Rietveld ASCII scan ( .RFL)
- GSAS ASCII scans ( .RAW), ( .XRA)
- ICDD PD3 ASCII scan ( .PD3)
- JCAMP ASCII scan ( .JDX or .JCP)
- LH-Rietveld ASCII scan ( .LHP)
- MDI ASCII scan ( .MDI)
- Rigaku ASCII scan ( .ASC), ( .FI),( .RAS), binary scan ( .RAW)
- Scintag binary scan ( .RAW)
- Seifert ASCII scan ( .ASC)
- Shimadzu binary scan ( .RAW)
- Skrowonek UDS2 ASCII scan ( .UDS)
- Stoe CSD-PROFAN ASCII scan ( .PRO),( .DAT), binary scan ( .RAW)
- Thermo Scientifique (ARL) scan ( .XML)
- ASD Inc. binary spectrum (.ASD)
Please note:
- HSvu requires high processing power and a large memory. The data file size handled by HSvu is limited by the device capabilities and its performance.
For a proper operation at least an A6 chipset, as present in the iPhone 5, is recommended. More recent devices with a 64-bit processor (like the A7 chipset) provide a very smooth user experience.
- To compare several scans you must insert them into a document in HSvu.
Скрыть
Показать больше...
Additionally, it shows and reports all details from an X-ray diffraction analysis performed by the HighScore analysis software from PANalytical (proprietary .HPF format).
Experience the power and ease-of-use of HighScore with HSvu:
- use six different graphical views for references, single and multiple scans, quantifications and 3-dimensional graphs
- pan and zoom-in for details like you do with HighScore
- get a complete overview on measurement conditions, scans, peaks, profiles and crystal structure data
- switch easily between graphics and textual information
- unravel the parameters used in a diffraction analysis and its history
- create reports covering either pieces or the complete diffraction document
HSvu is a display or viewing app.
It shows existing data, but it doesn't perform an analysis nor does it change data.
The following file formats are supported:
- PANalytical .HPF (proprietary HighScore document)
- PANalytical scans ( .XRDML)
- PANalytical ( .2Dint), ASCII format integrated from a 2D area detector
- ASD Inc. binary spectrum ( .ASD)
- Philips binary scans ( .RD), ( .SD), ( .FP), ASCCI ( .UDF), ASCII( .LIS)
- Bruker binary scans ( .RAW) versions 1, 2, 3 and 4
- ASCII scans ( .LST), ( .UXD), ( .XDD)
- Crystallographic information file: ( .CIF)
- General ASCII scan X-Y(-Z) table ( .ASC)
- DBW-Rietveld ASCII scan ( .RFL)
- GSAS ASCII scans ( .RAW), ( .XRA)
- ICDD PD3 ASCII scan ( .PD3)
- JCAMP ASCII scan ( .JDX or .JCP)
- LH-Rietveld ASCII scan ( .LHP)
- MDI ASCII scan ( .MDI)
- Rigaku ASCII scan ( .ASC), ( .FI),( .RAS), binary scan ( .RAW)
- Scintag binary scan ( .RAW)
- Seifert ASCII scan ( .ASC)
- Shimadzu binary scan ( .RAW)
- Skrowonek UDS2 ASCII scan ( .UDS)
- Stoe CSD-PROFAN ASCII scan ( .PRO),( .DAT), binary scan ( .RAW)
- Thermo Scientifique (ARL) scan ( .XML)
- ASD Inc. binary spectrum (.ASD)
Please note:
- HSvu requires high processing power and a large memory. The data file size handled by HSvu is limited by the device capabilities and its performance.
For a proper operation at least an A6 chipset, as present in the iPhone 5, is recommended. More recent devices with a 64-bit processor (like the A7 chipset) provide a very smooth user experience.
- To compare several scans you must insert them into a document in HSvu.
Скриншоты
HSvu Частые Вопросы
-
Приложение HSvu бесплатное?
Да, HSvu полностью бесплатное и не содержит встроенных покупок или подписок.
-
Является ли HSvu фейковым или мошенническим?
Недостаточно отзывов для надежной оценки. Приложению нужно больше отзывов пользователей.
Спасибо за ваш голос -
Сколько стоит HSvu?
Приложение HSvu бесплатное.
-
Сколько зарабатывает HSvu?
Чтобы получить оценку дохода приложения HSvu и другие данные AppStore, вы можете зарегистрироваться на платформе мобильной аналитики AppTail.

Оценки пользователей
Приложение еще не оценено в Тайвань.

История оценок
HSvu Отзывы Пользователей
Оценки

История позиций в топах
История рейтингов пока не доступна

Позиции в категории
Приложение еще не было в топах
HSvu Конкуренты
| Name | Скачивания (30d) | Ежемесячный доход | Отзывы | Оценки | Последнее обновление | |
|---|---|---|---|---|---|---|
|
Bruker SPR Tools
|
Открыть
|
Открыть
|
0
|
|
10 месяцев назад | |
|
單位轉換器 All-in-1
長度 面積 體積 質量 能量 功率 壓強 速度 時間 ...
|
Открыть
|
Открыть
|
0
|
|
7 месяцев назад | |
|
Daviz
|
Открыть
|
Открыть
|
0
|
|
3 года назад | |
|
splotRF
|
Открыть
|
Открыть
|
0
|
|
2 месяца назад | |
|
Molarity Calcs
Molarity Calculator
|
Открыть
|
Открыть
|
0
|
|
2 года назад | |
|
iMolecular Builder for iPad
|
Открыть
|
Открыть
|
0
|
|
3 года назад | |
|
Mapboard GIS
Draw your field maps into GIS
|
Открыть
|
Открыть
|
0
|
|
1 месяц назад | |
|
N/A
N/A
|
Открыть
|
Открыть
|
0
|
|
1 год назад | |
|
Stealth Creativity
|
Открыть
|
Открыть
|
0
|
|
8 месяцев назад | |
|
State Scan Lock
N/A
|
Открыть
|
Открыть
|
0
|
|
6 месяцев назад |
HSvu Установки
30дн.HSvu Доход
30дн.HSvu Доходы и Загрузки
Получите ценные инсайты о производительности HSvu с помощью нашей аналитики.
Зарегистрируйтесь сейчас, чтобы получить доступ к статистика загрузок и доходов и многому другому.
Зарегистрируйтесь сейчас, чтобы получить доступ к статистика загрузок и доходов и многому другому.
Информация о приложении
- Категория
- Productivity
- Разработчик
- Panalytical
- Языки
- English
- Последнее обновление
- 1.0.12 (1 год назад )
- Выпущено
- Aug 6, 2015 (9 лет назад )
- Также доступно в
- Индия , Китай , Нидерланды , Испания , Республика Корея , Соединенные Штаты , Бразилия , Турция , Румыния , Кувейт , Казахстан , Малайзия , Нигерия , Норвегия , Новая Зеландия , Филиппины , Польша , Португалия , Южно-Африканская Республика , Россия , Саудовская Аравия , Сингапур , Таиланд , Тайвань , Украина , Вьетнам , ОАЭ , Эквадор , Аргентина , Австралия , Бельгия , Беларусь , Канада , Чили , Чехия , Германия , Дания , Алжир , Япония , Египет , Финляндия , Франция , Великобритания , Греция , Гонконг (САР) , Индонезия , Ирландия , Израиль , Италия
- Обновлено
- 3 месяца назад
This page includes copyrighted content from third parties, shared solely for commentary and research in accordance with fair use under applicable copyright laws. All trademarks, including product, service, and company names or logos, remain the property of their respective owners. Their use here falls under nominative fair use as outlined by trademark laws and does not suggest any affiliation with or endorsement by the trademark holders.
AppTail.